Microscopie
Localisé au sein du Centre de Biologie et de Recherche en Santé, le plateau de microscopie en fluorescence de BISCEm est ouvert à l’ensemble des laboratoires de l’université de Limoges ainsi qu’aux structures extérieures, qu’elles soient publiques ou privées.
Il met à disposition des utilisateurs des systèmes de microscopie de fluorescence et d’analyse d’images en utilisation libre (après formation) ou assistée par l’ingénieur de la plateforme :
- 1 microscope en incubateur
- 1 microscope confocal
- 1 macroscope confocal
- 1 microscope plein champ
- 1 logiciel de déconvolution
- des logiciels d’analyse d’image
Personnel du plateau
Claire CARRION
Applications :
- Mosaïques d’images (pour acquisition de coupes complètes de tissus, par exemple)
- Acquisitions en z puis déconvolution des images avec le logiciel Huygens disponible sur la plateforme.
- Possibilité d’automatiser les acquisitions et d’associer les « dimensions » entre elles (fluorescences / multipositions /z stacking / mosaïques…) (Exemple : programmation d’acquisition de mosaïques sur des coupes de tissu à raison de 2 coupes par lame, possibilité de moduler les paramètres d’acquisitions en terme de nombre de fluorescences acquises et/ou temps d’exposition de la caméra en fonction des coupes…)
- ELISPOT sur plaque 96 puits et analyse du nombre et de la taille des spots.
Illustrations :
Applications :
- Acquisitions en 2D, 3D, 4D
- Mosaïques d’images en 2D et 3D
- Imagerie spectrale (acquisitions des spectres de fluorescence pour multiplexage et/ou identification des signaux de fluorescence endogène)
- Etude de colocalisation
Illustrations :
Equipement :
Types d’échantillons :Gros échantillons : petits modèles animaux (poisson zèbre, fourmi…), plantules, racines, coupes épaisse de tissus, céramiques, matériaux… |

Nikon AZ100 + tête CREST |
Applications :
- Acquisitions 2D et 3D
- Mosaïques d’images en 2D et 3D
Illustrations :
 |
 |
 |
 |
 |
|
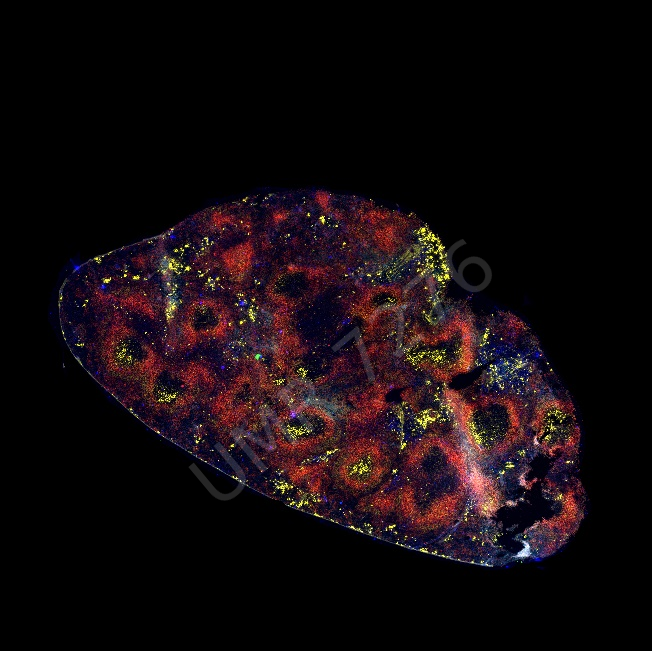
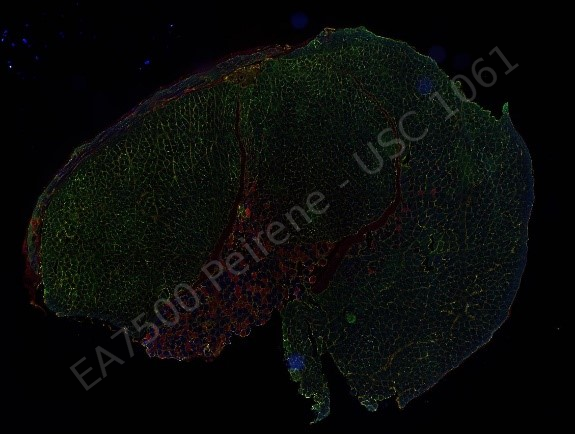
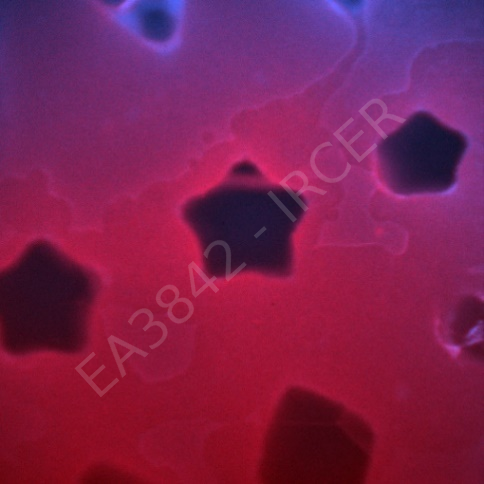
Peirene EA7500 Racine de tomate, Apex |
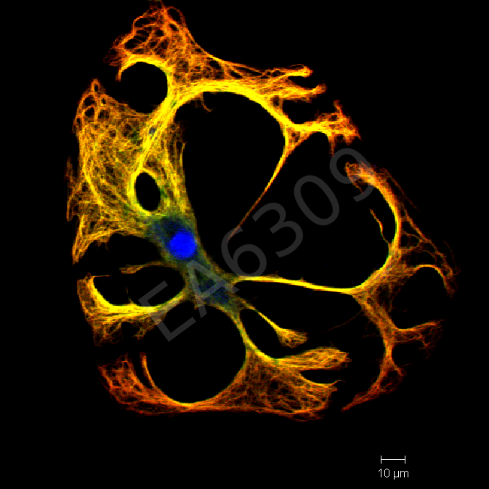
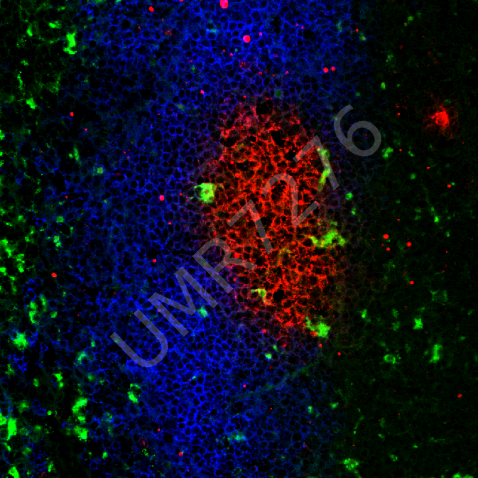
CAPTUR EA3842 / IRCER Cellules pré-ostéoblastiques murines marquées pour l’actine (rouge) et le noyau (bleu) cultivées sur pastille de céramique phosphocalcique fabriquée par microstéréolithographie. |
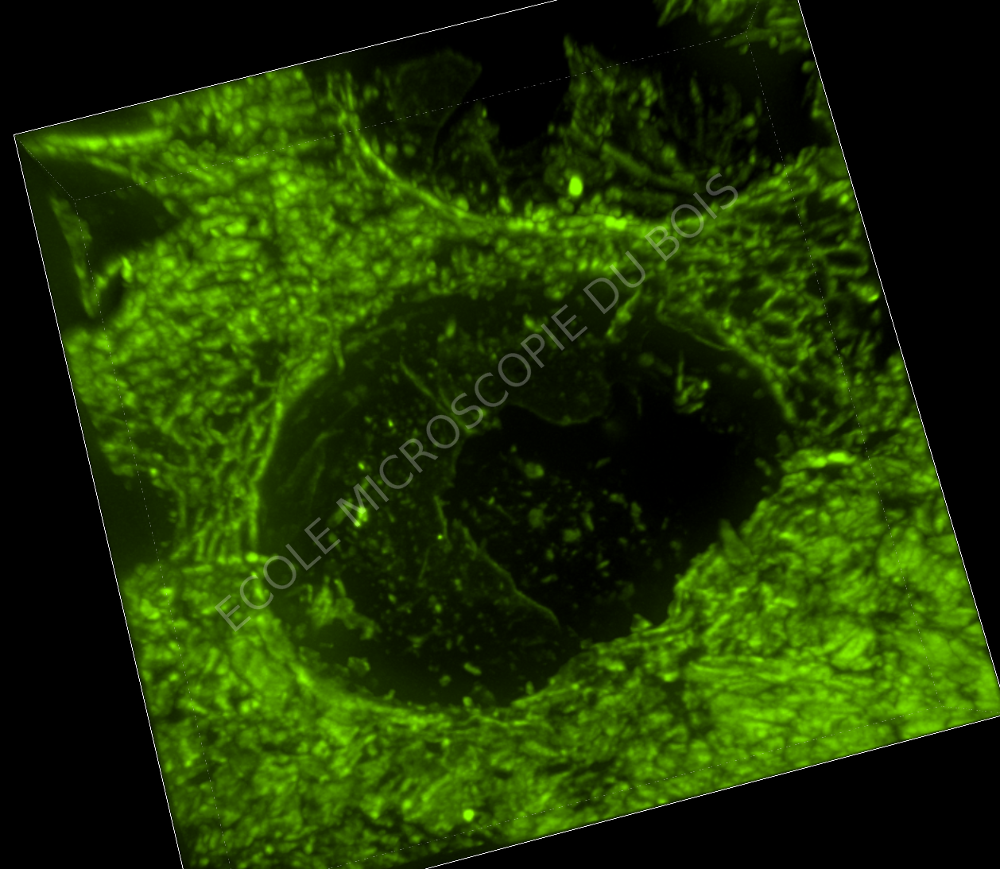
ECOLE DE MICROSCOPIE DU BOIS 2018 Boiserie en chêne et échantillon de bois |
||
Déconvolution des images : Logiciel Huygens
Applications :
Amélioration de la qualité des images obtenues avec les microscopes de fluorescence à partir du calcul de fonction d’étalement du signal (Point Spread Function) |
Illustration :F.I .S.H. Lymphocytes B activés |
Logiciels d’analyse d’images : Volocity, NiS-Elements AR, Image J, Imaris
Applications :
Quantifications en 2D (nombre d’objets, surface, distances, colocalisation…), 3D (nombre d’objets, volumes, distances, colocalisation, arborescences…), 4D (trajectoires, vitesse…) |
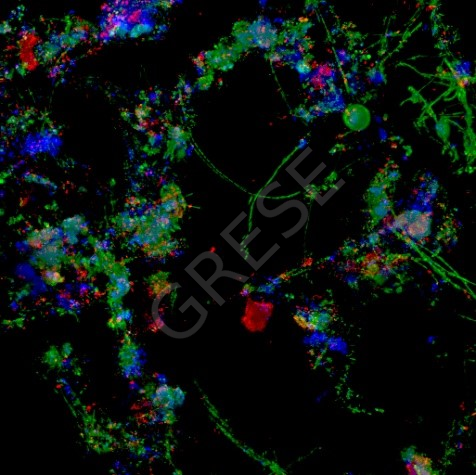
Illustration :RESINFIT UMR Inserm 1092 Analyse de la composition de biofilms bactériens |
Appareils accessibles en collaboration avec la plateforme PLATINOM (plateforme de l’Institut de Recherche XLIM UMR CNRS 7252 et service commun de l’Université de Limoges)
- Microscope Multiphotonique (Olympus)
Appareils accessibles en collaboration ou via des prestations avec la plateforme CarMaLim (plateforme du laboratoire IRCER UMR CNRS 7315 et service commun de l’Université de Limoges)
- Microscopes Electroniques (en transmission ou à balayage) : ZEISS Super Crossbeam FIB (en partenariat avec Xlim); Jeol 7400F (système cryogénique); FEI quanta 450 FEG ESEM
- Microscope à Force Atomique : Brucker Icon
- Spectroscope Raman 3D : Renishaw Invia Reflex
|
Microscopie multimodale (2PEF, 3PEF, SHG, THG, Raman, CARS, analyse spectrale haute résolution) En partenariat avec les plateformes PLATINOM et CARMALIM (pour toute demande concernant cet équipement : contact-imagerie@unilim.fr) |
 |
Modalités d'accès
N’hésitez pas à prendre contact avec le plateau (biscem.act@unilim.fr) pour plus d’information sur les conditions d’utilisation et les possibilités d’analyses que nous pouvons vous offrir.
Pour les utilisateurs autonomes, un calendrier de réservation est mis à disposition à cette adresse.
Pour toute demande ou réservation, un code projet vous sera demandé. Si vous n’en avez pas, merci de remplir le formulaire de dépôt de projet (utilisateurs de l’Université de Limoges uniquement). Pour les utilisateurs extérieurs, merci de prendre contact avec BISCEm pour l’attribution de votre code projet.